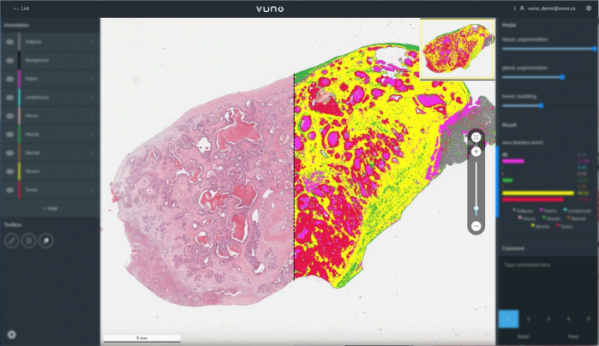
이번 공동연구는 딥러닝 알고리즘으로 암 조직 슬라이드 내 종양미세환경의 종양과 기질의 공간적 분포를 분석하고 암세포전이의 주요 원인이 되는 유전인자인 전령 RNA(messenger RNA, 이하 mRNA)의 발현율과의 연관성을 확인하기 위해 진행됐다.
연구팀은 뷰노의 인공지능 기반 병리 영상 정량화 및 분석 플랫폼인 뷰노메드 패스랩(VUNO Med®-PathLab)을 이용해 세계적인 암 유전체 데이터베이스(The Cancer Genome Atlas, TCGA)에서 대장암 환자의 암 조직 슬라이드를 분석했으며, 고가인 대장암 분석 방법 CMS 분석법과 유사한 대장암 조직 슬라이드 분석 결과가 도출될 수 있음을 확인했다. 이는 별도의 유전자 정보, 실험실 연구 등 추가적인 분석 없이, 조직 슬라이드만으로도 대장암 환자 치료에 필요한 정보를 RNA 기반의 전체 유전자 발현 분석 수준으로 확인할 수 있다는 점을 시사한다.
이에 연구팀은 더 나아가 대장암 조직 슬라이드의 기질 조직의 정량분석 결과가 종양을 유발하거나 억제할 수 있는 유전자와 연관이 있음을 밝혀냈다.
연구 결과에 따르면 대장암 내 기질 조직 비율이 높은 경우는 항원전달제시세포(APC), 자연살해세포(NK세포), 형질세포양 수지상세포(pDCs) 등 면역 관련 유전자의 발현이 낮고, 상피간엽이행,결합조직형성 반응, 섬유성아세포성 면역단백질과 같은 혈관 형성 관련 유전자 발현은 높았다.
MD앤더슨암센터 이선영 교수는 “해당 연구는 병리 슬라이드만으로도 자동으로 조직을 분할(Segmentation)하고 유전자 정보를 함께 분석하는 딥러닝 기반 분석법의 가능성을 확인했다는 점에서 의미가 있다”며, “후속 연구와 검증을 통해 기존 대장암 조직 분자 유전 검사 대비 비용효과적인 대장암 진단 및 치료 바이오마커로 활용될 수 있을 것”이라고 말했다. 또한 “향후 후속 연구를 통해 대장암뿐만 아니라 다른 암종에서도 발병 예측과 환자의 치료 방향성 결정에도 기여할 수 있을 것으로 기대한다”고 덧붙였다.

!["이미 결제된 선수입니다"…본격 막 올린 스토브리그, '변수'는 없을까? [이슈크래커]](https://img.etoday.co.kr/crop/140/88/2098040.jpg)
![먹을 사람 없고 귀찮아서…"김장, 안 하고 사 먹어요" [데이터클립]](https://img.etoday.co.kr/crop/140/88/2098061.jpg)

![[단독] "AI에 진심" 정태영 부회장, '유니버스' 중동 세일즈 나섰다](https://img.etoday.co.kr/crop/140/88/1970536.jpg)







![[내일 날씨] 전국 대부분 아침 기온 5도 내외…얼음 어는 곳도](https://img.etoday.co.kr/crop/85/60/2098154.jpg)







![[정치대학] 美 대선, 막판까지 초박빙…당선자별 韓 영향은?](https://img.etoday.co.kr/crop/300/170/2098110.jpg)
!['한국의 장 담그기 문화' 유네스코 무형문화유산 등재 유력 [포토]](https://img.etoday.co.kr/crop/300/190/2098066.jpg)